本站讯(通讯员 高峰)近日,天津大学生物信息中心高峰教授课题组与英国Newcastle University的Heath Murray教授课题组合作,在细菌复制起始区解旋的基本机制研究方面取得新进展。
在模式生物枯草芽孢杆菌的研究中,Heath Murray教授领导的研究小组发现,位于染色体复制起始点解旋区内的重复性三核苷酸基序(DnaA-trios)是复制起始蛋白DnaA打开DNA双螺旋所必需的元件(如A图所示)。该研究组的实验结果表明,DnaA蛋白首先通过特定序列DnaA盒(DnaA-box)结合双链DNA,然后引导其他的DnaA单体与DnaA-trios结合,形成DnaA细丝,从而促进DNA双链的打开(Richardson et al., Nature, 2016; Richardson et al., EMBO J, 2019)(如B图所示)。然而,以上对枯草芽孢杆菌复制起始区解旋机制的描述,是否代表了不同细菌物种之间共有的保守性机制成为研究的焦点问题。在Murray教授的邀请下,高峰教授课题组采用生物信息学的方法,寻找不同细菌染色体复制起始点序列中是否存在类似的基本解旋系统(Basal Unwinding System,缩写BUS)。生物信息学预测结果表明,在整个细菌域的复制起始点中广泛存在着可能的BUS序列元件(约占所研究物种的85%)(如C图所示)。在此基础上,Murray教授研究组先后测试了不同革兰氏阳性菌和阴性菌的DnaA同源物在体外解旋相应复制起始区的能力,发现DnaA活性取决于基本解旋系统BUS(包括DnaA-box和DnaA-trios)的存在,表明DNA双链分离的机制很可能是保守的。幽门螺杆菌体内实验结果同样表明,基本解旋系统对该菌的生存不可或缺。
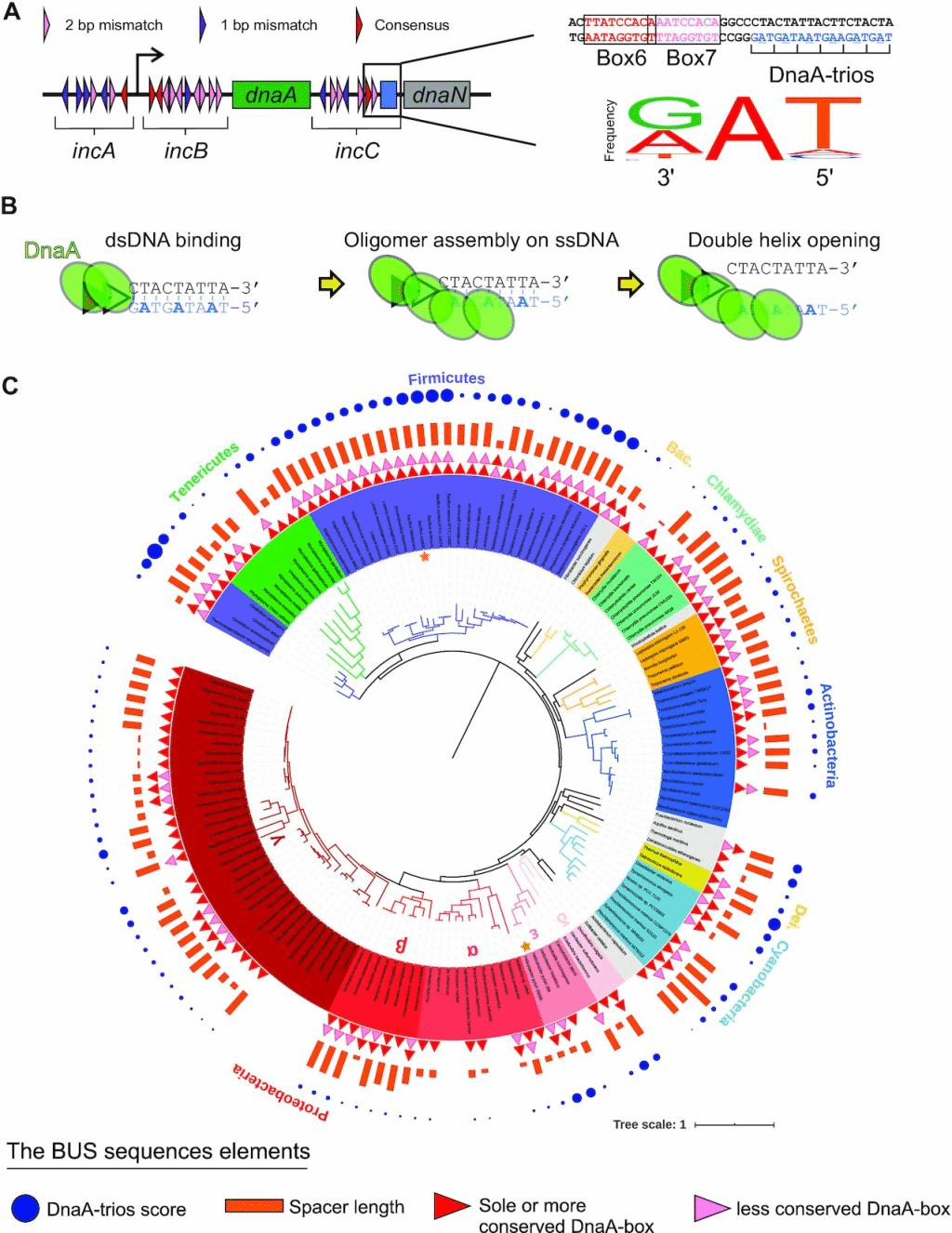
生物信息学分析识别出整个细菌域复制起始区内可能的BUS序列元件
这项工作将极大地促进我们对复制起始区结构和DNA双链解旋机制的基本理解,从而建立细菌DNA复制起始机制的一般性描述。以上研究发表在生物信息学领域顶尖期刊《Nucleic Acid Research》(中科院分区一区Top期刊,最新影响因子16.971)上,论文题目为“Evidence for a chromosome origin unwinding system broadly conserved in bacteria”(论文链接https://doi.org/10.1093/nar/gkab560)。高峰教授为本文的共同通讯作者,指导理学院博士生董美敬(本文第二作者)完成了论文生物信息学预测算法的设计和实现。
年初,高峰教授课题组还和美国Wayne State University张任教授合作,以共同通讯作者身份在《Nucleic Acids Research》发表新版本必需基因数据库DEG 15的论文(论文链接 https://doi.org/10.1093/nar/gkaa917)。理学院青年教师罗昊博士为论文的第一作者(Luo et al., Nucleic Acid Research, 2021)。DEG数据库由天津大学生物信息中心维护,2003年起对外提供服务,目前已成为必需基因研究领域必不可少的数据库,累计SCI引用900余次。
(编辑 焦德芳 张青林)